
NadPrep®️ 血浆游离DNA双端分子标签文库构建试剂盒(for Illumina®️)
该试剂盒搭配双端分子标签(Unique Molecular Identifier, UMI)接头,可满足血浆游离 DNA 超低频突变分析的需求。
- 产品详情
- 产品组分
- 常见问题
- 订购信息
- 资料下载
产品特色
- 适用于血浆游离 DNA
- 样本起始量低,文库转化率高
- 双端分子标签接头助力低频突变分析
- 包含 DNA 纯化磁珠,并适配优化的片段筛选方案
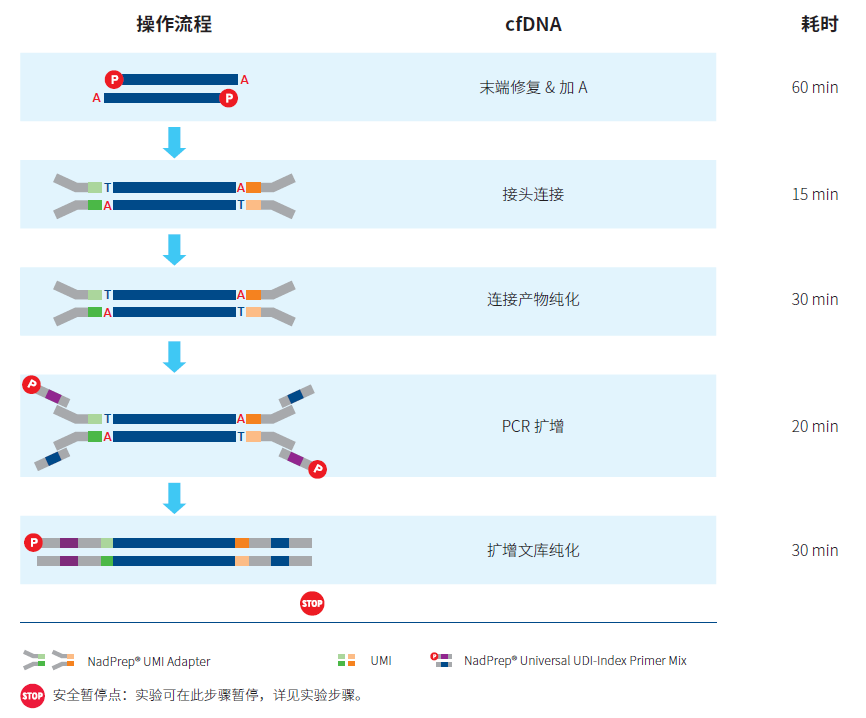
建库流程
产品表现
来源于 2 个健康捐献者的血浆游离 DNA 以 1% 比例混合,用于模拟游离血浆 DNA 中低频突变位点的检测,文库制备总投入量为 10 ng;针对已知 SNP 位点设计捕获 panel(图 1)。通过 Illumina HiSeq X, PE150 平台测序后,利用双端分子标签(UMI),在不同分析与过滤方法下进行低频突变分析(表 1)。

图 1. 低频突变模拟模型。来源于2个健康捐献者的 cfDNA 以 1% 比例混合,文库制备总投入量为 10 ng。1% 及 0.5% 模拟突变位点数量统计如右表所示。
表 1. 分析与过滤方法
|
分析模式 |
详情 |
|
No UMI |
根据片段在基因组上比对的起始、终止位点去除重复。 |
|
SSCS |
利用单链分子标签进行一致性分析(Call Molecular Consensus Reads),形成单链一致性序列(Single Strand Consensus Sequences, SSCS)。 |
|
DCS211 |
利用双链分子标签,即互补链(Top / Bottom)分子标签进行一致性分析,形成双链一致性序列(Duplex Consensus Sequences, DCS),且Top与Bottom Reads均 ≥ 1。 |
|
DCS633 |
利用双链分子标签,即互补链(Top / Bottom)分子标签进行一致性分析,形成 DCS,且 Top 与 Bottom Reads 均 ≥ 3。 |
|
DCS211 (≥ 2) |
在 DCS211 的基础上,支持该突变的 DCS 一致性序列 ≥ 2。 |
|
DCS633 (≥ 2) |
在 DCS633 的基础上,支持该突变的 DCS 一致性序列 ≥ 2。 |
突变频率符合预期

图 2. 1% 与 0.5% 模拟突变位点的频率估算。
不同分析模式过滤背景“噪音”

图 3. 不同分析模式对背景噪音的过滤。
灵敏度与阳性预测值

图 4. 1% 和 0.5% 模拟突变位点在不同分析模式下的灵敏度(A)与阳性预测值(B)。
本产品仅限研究使用,不用于临床诊断。
通用建库模块
/
/
包装编号
管盖颜色
组分名称
包装体积
1002101 (24 rxn)
包装体积
1002103 (96 rxn)
Box1
EndRepair & A-Tailing Buffer
180 µL
690 µL
End Repair & A-Tailing Enzyme
120 µL
460 µL
Ligation Buffer
750 µL
2×1500 µL
DNA Ligase
65 µL
250 µL
2X HiFi PCR Master Mix
720 µL
2×1600 µL
NadPrep
Amplification Primer Mix II
145 µL
650 µL
Nuclease Free Water
2.5 mL
8 mL
TE Solution
1 mL
4 mL
Box2
NadPrep SP Beads
4 mL
15 mL
分子标签接头模块
管盖颜色
组分名称
包装体积
1103111 (24 rxn)
包装体积
1103121 (96 rxn)
包装体积
1103122 (96 rxn)
NadPrep Universal UDI-Index
Primer Mix #
12×15 µL
24×25 µL
24×25 µL
NadPrep UMI Adapter
60 µL
230 µL
230 µL
NadPrep Amplification Primer Mix II
20 μL
75 μL
75 μL
NadPrep® 血浆游离 DNA 双端分子标签文库构建试剂盒对 cfDNA 样本的定量及片段质检,建议如下:
|
类别 |
要求 |
||
|---|---|---|---|
|
提取试剂盒推荐 |
cfDNA |
QIAamp Circulating Nucleic Acid Kit(Qiagen;货号:55114) |
|
|
溶剂 |
推荐使用无核酸酶水、0.1 × TE(low TE)、10 mM Tris-HCl(pH 8.0~8.5)或提取试剂盒配备的Buffer溶解DNA样品。酶切片段化试剂多对EDTA浓度敏感,应避免使用含EDTA的溶剂溶解。如有必要可进行1.8-2.0 ×磁珠纯化去除。 |
||
|
定量 |
基于分光光度计原理的NanoDrop和基于双链DNA荧光染料的Qubit®、PicoGreen®进行定量。 |
||
|
纯度 |
OD260/OD280=1.8-2.0;OD260/OD230=2.0-2.5。OD260/OD280>1.9表明有 RNA 污染,<1.6表明有蛋白质、酚等污染;OD260/OD230<2.0,则表明有碳水化合物、盐类或者有机溶剂污染。 |
||
|
片段分布 |
cfDNA |
Bioanalyzer或Bioptic(Qsep)分析主峰~160 bp,次峰~320 bp。 |
|
纳昂达针对 Illumina® 平台目前有以下接头类型可供选择,用以与 NadPrep® DNA建库模块搭配,形成完整的 DNA 文库构建体系。
| 接头类型 | 说明 | 推荐应用 | |||||||||||||||||||||||||||||||||
| NadPrep® UDI Adapter | 双端唯一 8nt index 全长接头 | 全基因组,宏基因组,液相杂交靶向捕获测序 | |||||||||||||||||||||||||||||||||
| NadPrep® Universal Stubby Adapter | 双端唯一 10 nt index,当文库在 Illumina 平台测序时,支持双端 8 nt 或 10 nt 的标签读取模式,1-384 种 | ||||||||||||||||||||||||||||||||||
| NadPrep® UMI Adapter | 含随机分子标签通用短接头,可搭配双端唯一 10 nt index 引物,构建全长文库,1-48 种 | 低频突变检测(~1% 或以下突变频率) | |||||||||||||||||||||||||||||||||
接头比例过高会导致接头或接头二聚体残留;接头比例若不足则会影响连接效率进而降低文库产量。接头使用还应注意保存方式、稀释方法和稀释浓度:
• 保存:避免反复冻融,如欲多次使用,应提前分装;
• 稀释液:推荐使用试剂盒内TE Solution进行接头的稀释,但稀释液不建议长期保存;
• 稀释用量:根据样本起始投入量、片段大小,计算合适的接头与DNA片段摩尔比,可达最佳产量。具体推荐摩尔比,可参考相关说明书。
• 实验操作:保持连接反应在冰上进行,接头与连接体系分别加入,避免提前自连。
文库长度分布可通过凝胶电泳、Bioanalyzer或者 Bioptic(Qsep)等生物分析系统进行检测,但应注意不同分析仪器的文库片段读数可能会存在一定的偏差。常见的异常文库片段分布原因如下:
|
异常峰型 |
产生原因 |
解决办法 |
|---|---|---|
|
有短片段(~100 bp)小峰 |
接头冗余 |
合适的Input DNA量和接头使用量;注意加样及连接条件 |
|
有双峰(额外单个短片段峰) |
过度扩增 |
降低Input DNA量,降低扩增循环数 |
|
额外有长片段峰 |
磁珠残留 |
纯化步骤时小心操作,以去除全部磁珠 |
|
产品 |
详情 |
货号 |
|
NadPrep®️ DNA Library Preparation Kit (for Illumina®️) G24 |
24 rxn |
1002101 |
|
NadPrep®️ DNA Library Preparation Kit (for Illumina®️) E96 |
96 rxn |
1002103 |
|
NadPrep®️ UMI Adapter Kit Set A1 (with 10 nt Index), 24 rxn |
1-12 |
1103111 |
|
NadPrep®️ UMI Adapter Kit Set B1 (with 10 nt Index), 96 rxn |
1-24 |
1103121 |
|
NadPrep®️ UMI Adapter Kit Set B2 (with 10 nt Index), 96 rxn |
25-48 |
1103122 |
本产品仅限研究使用,不用于临床诊断。
我们的工作人员将在1个工作日内与您取得联系。
如果有任何问题,欢迎联系
400 8717 699 或 support@njnad.com。











